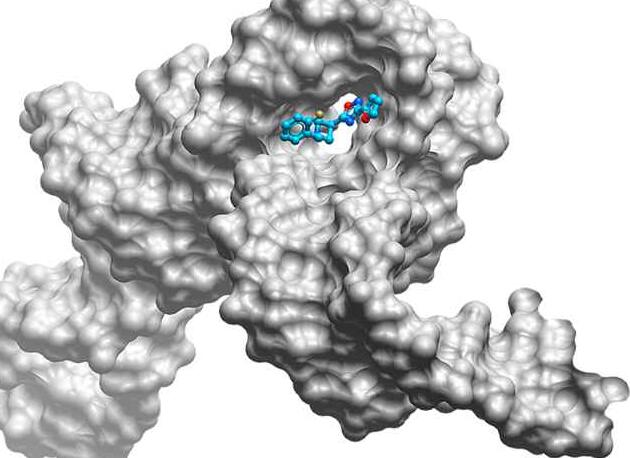
冷冻电子显微镜现在是确定蛋白质结构的最流行的方法,这有助于研究人员为不同种类的疾病开发药物。在过去的几十年里,它已经取代了X射线晶体学,因为它可以成像不易形成大晶体的蛋白质。这项新技术非常具有革命性,它为开发商赢得了2017年诺贝尔化学奖。
cryo-EM的最终产品是生物分子中原子密度的图,但为了达到研究人员所需的细节水平,他们需要进行进一步的分析。“自然方法”杂志上的一项新研究概述了一种将低分辨率地图提高到标准的技术。
研究人员用来做这个的方法取决于他们开始的细节水平。2至3ångström(Å,用于表示原子和分子大小的长度单位)的地图通常被认为是高分辨率的。然而,这种质量的图很难实现,并且许多仍然通常在4到10埃的范围内生产。在2016-18期间沉积到电子显微镜数据库的所有蛋白质中,超过50%以中等分辨率解析。
“如果分辨率优于3,那么常规工具可以追踪氨基酸位置并建立原子位置图。但是经常冷冻EM不能给你3Å地图,”生物科学和计算机教授Daisuke Kihara说。普渡大学的科学。“在5Å或更低的地图中,你通常根本看不到链条连接。”
蛋白质实际上是氨基酸链,氨基和羧基之间的键合有时会产生某些折叠模式。这些模式称为α螺旋和β链,形成蛋白质的二级结构。
在5到8Å的地图中,蛋白质二级结构的一些片段通常是可见的,但追踪整个链条将非常困难。Kihara的新方法,即Emap2sec,揭示了地图中从6到10的二级结构。
Emap2sec在其算法的核心有一个深度卷积神经网络。这些网络是深度学习系统,主要用于对图像进行分类,通过相似性对它们进行聚类并执行对象识别。它适用于3D地图中的蛋白质结构识别,因为当信息通过神经网络层时,该方法将局部地图密度特征“卷积”到更大区域的图像。局部预测是在地图的大区域的背景下进行的。
三维地图中识别出的二级结构有助于研究人员将已经解析过的已知蛋白质结构分配到地图中。这意味着他们有时会有一个起点,或者至少是一些结构看起来像什么的线索。Emap2sec可以帮助研究人员更快速,更轻松地将他们的作品融入到拼图中。已识别的结构信息也可能有助于在结构建模中发现错误。
该程序现已在软件开发平台GitHub上提供。该研究得到了美国国立卫生研究院,国家科学基金会和普渡药物研究所的支持。